Hola amantes del tocino. Esta semana tenemos un post invitado en el blog. El autor es Tomás Corrales, doctor en Física de la Universidad de Mainz, Alemania. Él nos cuenta sobre los temas que lo matuvieron ocupado en los últimos años, durante su doctorado y de su actual investigación sobre ADN, que lleva a cabo como investigador postdoctoral en la Universidad de Cornell, en EEUU.
¿Se
han preguntado alguna vez de que están hechos todos estos materiales
relativamente blandos que se encuentran a nuestro alrededor y que
usamos día a día? Sólo por nombrar algunos ejemplos: las bolsas del
supermercado, los envases de yogurt, los recubrimientos de las
píldoras, las llantas de un auto y los pañales para bebés. El
denominador común entre todos materiales es que están hechos de
moléculas muy especiales llamadas macromoléculas. Las
macromoléculas se encuentran en casi todos los materiales que usamos
a diario, (ver
más ejemplos), pero aún más importante, son fundamentales en
la organización de la vida como la conocemos. Dos grandes ejemplos
biológicos de macromoléculas son las proteínas y nuestro propio
ADN, en cuya estructura se codifica la información genética de los seres vivos.
¿Pero,
qué es una macromolécula? La palabra en sí lo dice todo, es una
molécula…“gigante”, tan grande, que agregarle o quitarle unos
cuantos átomos no afectan sus propiedades, es casi como si le
sobrasen átomos. Típicamente una macromolécula puede tener
dimensiones desde 1 nanómetro (la millonésima parte de un
milímetro) hasta llegar a los micrones (la milésima parte de un
milímetro).
Si
bien hoy en día el uso de macromoléculas es trivial, y cada día
aparecen productos con propiedades mejoradas (más ligero, más
resistente, etc), su descubrimiento y la manipulación de sus
propiedades no lo fueron tanto. Materiales hechos de macromoléculas,
como el caucho, las fibras de algodón e incluso los primeros
plásticos, ya eran utilizados mucho antes de que se supiese
realmente de que estaban hechos. No fue sino hasta 1920 que el
concepto de macromolécula fue propuesto por el químico alemán Hermann Staudinger. En un comienzo, la idea de Staudinger fue
controversial. Los científicos de la época creían que materiales
como el caucho consistían en grandes agrupaciones de moléculas
pequeñas que eran atraídas por fuerzas eléctricas, formando
partículas o coloides. Staudinger en cambio, fue el primero que
propuso que materiales como el caucho estaban hechos de
macromoléculas compuestas por cientos o miles de átomos unidos por
enlaces covalentes. Al cabo del tiempo, los desarrollos
experimentales y teóricos de la década de los 20 le dieron la razón
a Staudinger, y de paso, el premio Nobel de química en 1953. En la
naturaleza se pueden encontrar muchos tipos de macromoléculas
distintas, pero en el tocino de hoy sólo hablaré de dos, polímeros
sintéticos y el ADN.
Los
plásticos son un ejemplo de materiales que están hechos de
polímeros sintéticos. Un polímero se caracteriza por ser una
especie de culebra (figura 1a), con una unidad química que se repite
a lo largo del espacio, formando una cadena. Uno de los polímeros más
simples es el polietileno (figura 1b), el cual consiste en átomos de
carbón unidos por enlace covalentes formando una cadena lineal. A
los costados de esta cadena se conectan además átomos de hidrógeno.
En forma condensada, los polímeros pueden formar materiales con
distintas propiedades. El largo de la cadena, sus ramificaciones y la
flexibilidad intrínseca de la molécula controlan la dureza del
plástico y también a qué temperatura se ablanda. Si hiciéramos un
zoom a una bolsa de plástico de polietileno y tomásemos un área de
10x10 nanómetros, veríamos una especie de tallarinata de moléculas
(figura 2).
Para
no aburrirlos con definiciones tediosas, pasemos a algo un poco más
entretenido, ¿Cómo estudiar y visualizar estas macromoléculas? ¿O
más interesante aún, como estirar o apretar macromoléculas
individuales? Sin duda, uno de los instrumentos más versátiles y
revolucionarios en nanotecnología ha sido el microscopio de fuerza
atómica (o AFM por sus siglas en inglés). Este instrumento permite
visualizar, así como también manipular macromoléculas individuales. El
AFM fue desarrollado en 1989 por Gerd Binning y Heinrich Rohrer, sólo
unos cuantos años después de la invención del microscopio de
efecto túnel (STM), por el cual Binning y Rohrer recibieron el
premio Nobel de física de 1986. El AFM funciona palpando el objeto
o superficie, utilizando una aguja de silicio del tamaño de algunos
átomos; digamos que éste es como un microscopio ciego (figura 3a).
Esta técnica ha permitido a científicos ver objetos más chicos que
cualquier método óptico haya podido resolver, llegando a ver átomos
individuales. La aguja atómica de un AFM se sitúa al final de una
barrita de silicio (figura 3b y c), la cual se deforma cada vez que
la punta de la aguja toca la superficie. Al barrer esta aguja de
silicio sobre la superficie un objeto se genera un mapa de la
deformación de la barra de silicio, la cual nos da información
sobre la topografía de nuestra superficie. El AFM puede hacer
imágenes de casi cualquier material, sea aislante o conductor, lo
cual le da una ventaja por sobre el STM que requiere de superficies
conductoras.
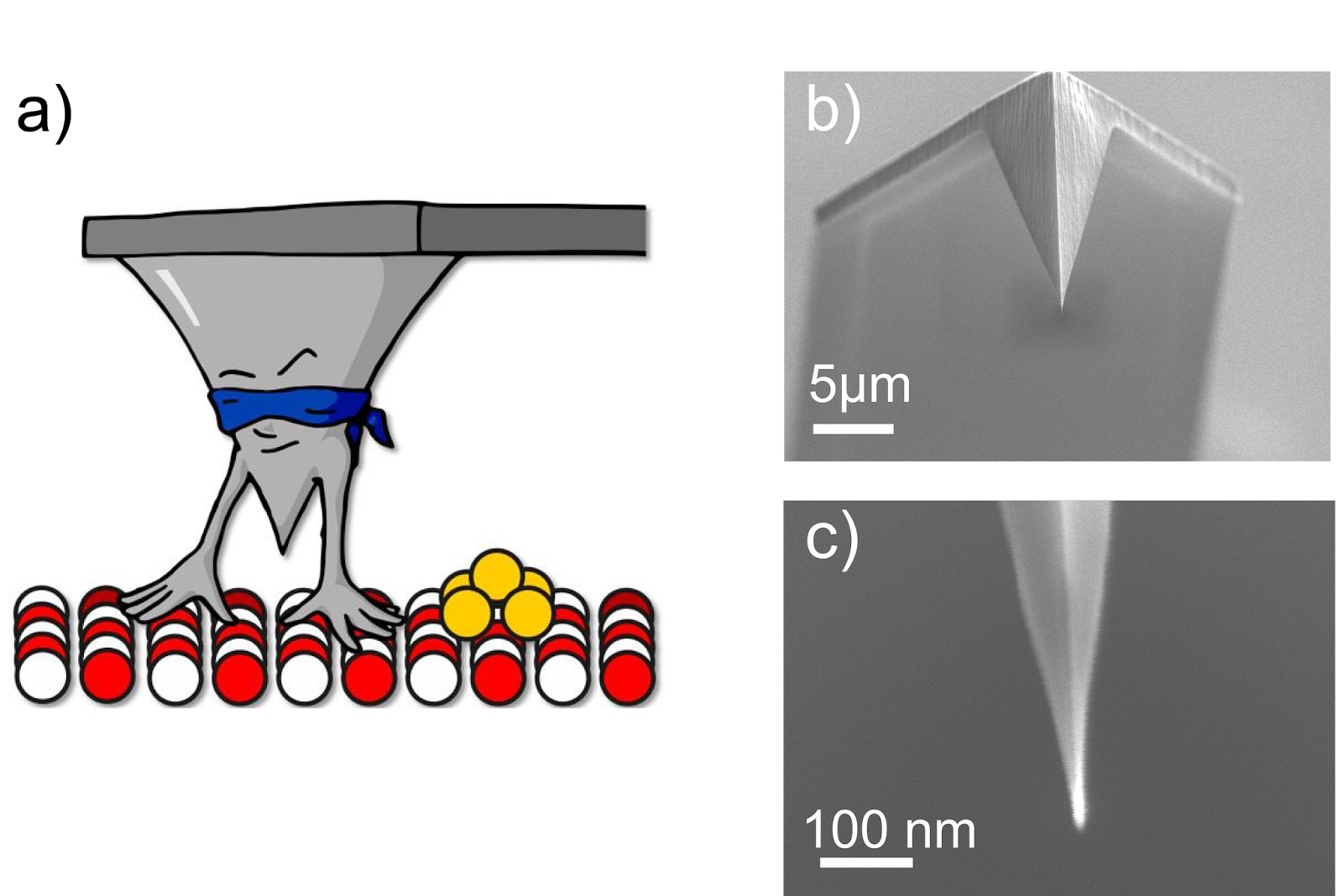 |
Figura
3: a) Caricatura del AFM, un microscopio ciego que “siente” los
átomos de una superficie (círculos rojo y blancos). Ojo, la punta
del AFM no tiene manos, lo que siente la aguja del AFM son fuerzas de
repulsión y atracción eléctricas entre los átomos de la punta y
los átomos de la superficie. Las imagen b) muestran una punta de AFM
real con su respectiva barrita de silicio (cantiléver en inglés).
En c) se muestra una imagen magnificada de la punta. La punta de una
aguja comercial puede llegar a tener entre 1 y 5 nanometros. Las
imágenes b) y c) fueron obtenidas usando un microscopio de barrido
electrónico (SEM por sus siglas en inglés) y se pueden encontrar en
mi tesis :) (Link).
|
Otra ventaja del AFM es que se puede utilizar en
distintos medios, por ejemplo, en medios acuosos, los cuales son
relevantes para estudiar macromoléculas biológicas. Un ejemplo de
la capacidad de esta técnica que me parece interesante, fue
publicado hace alrededor de 2 años, en donde se visualiza por
primera vez en espacio real la doble hélice de una molécula de ADN.
La estructura de doble hélice del ADN fue descubierta hace más de
50 años usando difracción de rayos X (esto da para otro tocinazo),
pero hasta ahora nadie había generado una visualización es espacio
real de esta hélice. En la imagen generada por AFM aparece una
molécula individual de ADN, si, es ese tallarín largo que aparece
acostado sobre la superficie plana (figura 4a)!. La doble hélice
aparecen como líneas diagonales al eje largo de la molécula,
indicadas con flechas verdes (figura 4b).
 |
Figura
4: a) Una molécula individual de ADN acostada sobre una superficie
plana. La imagen fue tomada en un medio líquido. La región
encerrada en el cuadrado punteado indica donde se hará una
magnificación. b) Imagen magnificada tomada de la imagen anterior.
En esta imagen se puede ver la doble hélice del ADN indicado por
pares de flechas verdes. Estas imágenes fueron obtenidas por el
grupo del Dr. Bart Hoogenboom, investigador del University Collegue
London, y aparecieron publicadas hace dos años en Nano
Letters, 2012, 12 (7),
pp 3846–3850. Este año este grupo ha obtenido imágenes aún más
espectaculares de la doble hélice (link).
|
Aparte
de poder visualizar macromoléculas ocupando AFM, se ha demostrado
que se pueden medir las propiedades mecánicas de macromoléculas
individuales usando la punta del AFM para estirarlas. La fuerza con
la que se pueden estirar una macromolécula ocupando AFM va desde los
cientos de nanonewton (1 nN = 0.000000001 N) hasta la aproximadamente
la centésima parte de un nanonewton (0.01 nN). Para poner esto en
contexto, si tomase una manzana de alrededor de 100 gramos, esta
ejercería una fuerza de 1 Newton sobre mí mano. Usando estas
pequeñísimas fuerzas se pueden estirar varios tipos de
macromolécula, incluyendo proteínas, polímeros y ADN. Una
aplicación de esto es estudiar cómo se doblan las proteínas.
Desafortunadamente las fuerzas involucradas en procesos biológicos
relevantes para el ADN son mucho menores a las fuerzas que se pueden
lograr utilizando un AFM. Típicamente para leer y copiar un segmento
de ADN, proceso que ocurre constantemente dentro de nuestro cuerpo,
se requieren del orden de milésimas de nanonewton (0.001 nN = 1
piconewton - pN). Tales fuerzas están fuera del alcance de un AFM,
por lo cual nuevas técnicas han sido desarrolladas. Una de estas
técnicas, que ha tomado relevancia en las últimas décadas para
aplicar fuerzas del orden de piconewtons a macromoléculas, son las
llamadas pinzas ópticas. Utilizando pinzas ópticas, se pueden
estirar moléculas como el ADN con fuerzas iguales o menores a 1 pN
(= 0.000000000001 Newtons, y si, hay 12 posiciones decimales en
ese número). Las pinzas ópticas funcionan bajo el siguiente
concepto básico: atrapar y manipular micropartículas que se
encuentran flotando en una solución liquida. Tales micropartículas
pueden ser, por ejemplo, esferas de vidrio del tamaño de 1
micrómetro (milésima de un milímetro). En el caso de las pinzas
ópticas, la microparticula se atrapa usando la radiación óptica de
un láser. La microparticula es atraída y queda atrapada cerca del
punto donde el láser se enfoca. Al mover la posición del foco del
láser lateralmente dentro de la solución, uno puede mover la
micropartícula (figura 5a). Ahora imaginen que esta microparticula
está anclada a una superficie por medio de una molécula de ADN, tal
como un niño que sujeta un globo de helio con un cordel (figura 5b).
Al mover la microparticula, el ADN se estira y se genera una fuerza.
Esta trampa óptica puede estirar un pedazo de ADN con fuerzas que
van desde los 0.1 pN hasta ~50 pN. Si uno grafica la fuerza con que
se estira el ADN versus su extensión (figura 5c) uno se da cuenta
que no se comporta como un elástico. Según la ley de Hooke un
elástico se comportaría de manera lineal en un gráfico de fuerza
versus extensión. El ADN en este caso tiene dos contribuciones, a
bajas fuerzas (menos a 20 pN), el ADN se tiene que desenredar de su
estado relajado (y desordenado). Cuando ya totalmente estirado (sobre
20 pN) el ADN empieza a comportarse como un elástico.
Las
pinzas ópticas permiten estudiar procesos biológicos, como la
transcripción de segmentos de ADN, replicando las condiciones en las
que suceden dentro del cuerpo humano, lo cual era impensado hace tan
sólo 20 años. Como si fuese poco, en los últimos años, científicos
han desarrollado formas de rotar micropartículas para así aplicar
torque sobre moléculas individuales de ADN.
¿Cuál
es el futuro de todo esto? Esta es una pregunta personal, que no
tiene una respuesta única, sino más bien una humilde opinión. Para
mí, uno de los mayores retos en esta área es entender que rol
juegan las propiedades mecánicas del ADN al momento de enrollarse
para formar el cromosoma. El cromosoma humano suele tener un tamaño
del orden de micrómetros, si desenrollásemos esta estructura
obtendríamos una fibra de ADN que llegaría a tener centímetros de
largo. El proceso de densificación del ADN para llegar formar el
cromosoma es bastante complejo (figura 6), entender como el ADN se
dobla y tuerce nos dará un entendimiento más profundo de este
proceso. Por otro lado, sería interesante estudiar como un pedazo
individual de ADN interactúa con la masa densificada de ADN que es
el cromosoma. ¿Existirán secuencias de ADN que son más flexibles
que otras? ¿Sufrirá algún cambio estructural el ADN al enrollarse
en el cromosoma? Estas son solo algunas de las preguntas que me
motivan a estudiar estos bichos, además de la posibilidad de
desarrollar nuevas técnicas con las cuales estirar y jugar con estas
macromoléculas, molécula a la vez.
 |
Figura
6: Los distintos niveles de compactación del ADN para formar el
cromosoma humano.
|



Muy interesante el artículo Dr. Corrales
ResponderBorrarjajajaja, muchisimas gracias Dr. Salvo
Borrar